<h3 style="text-align:center;">R - aplicado a estudios herpetológicos</h3> <h3 style="text-align:center;">Clase 2: Trabajando con datos</h3> <p style="text-align:center;">Fernanda R. de Avila y Raúl Maneyro</p> <p style="text-align:center;">fernandar.avila@gmail.com</p> <p style="text-align:center;">Lab. de Herpetología - FCIEN, UDELAR</p> <img src="index_files/figures/cry.png" width="30%" style="display: block; margin: auto;" /> --- layout: false class: inverse, middle, center # Trabajando con datos --- ## Sobre el material de la clase Ahora que ya has dado tus primeros pasos en el ambiente R, vamos a avanzar un poco. En esta etapa vamos a aprender: `\(~\)` - Configurar y guardar un proyecto; - Configurar el directorio de trabajo; - Insertar datos externos en el ambiente de trabajo; - Trabajar con diferentes fuentes de datos en R; - Exportar los resultados de nuestro trabajo de R a la memoria de la máquina; - Buenas prácticas escribiendo comandos. --- layout: false class: inverse, middle, center # Configurar un proyecto --- ## Configurar un proyecto .center[ Guardar un proyecto = **organización, reproducibilidad y eficiencia** Archivo **.Rproj** guardar tus análisis y toda su información (datos, scripts y outputs) ] #### Interesante para: - Trabajos largos, con muchos análisis - Trabajar en etapas sin perder tu progreso - Ejemplo: bioinformática o modelado de distribución geográfica `\(~\)` .center[ #### Para guardar un proyecto, en RStudio, sigue los siguientes pasos: `File > New Project > New Directory > New Project` ] --- layout: false class: inverse, middle, center # Directorio de trabajo --- ## Directorio de trabajo `\(~\)` `\(~\)` `\(~\)` .center[ R asume que los archivos que vas a utilizar en tus análisis están todos almacenados en **la carpeta donde el propio R fue instalado** ] --- ## Organizar tu trabajo en R Reunir todos los archivos que deseas trabajar en una carpeta en tu computadora y definir esa carpeta como tu directorio de trabajo. `\(~\)` #### Carpetas estructuradas ``` cursoR/ ├── cursoR.Rproj │ ├── 00_scripts/ │ ├── 01-carregar-datos.R │ ├── 02-analise.R │ └── 03-graficos.R │ ├── 01_datos/ │ └── datos.csv │ └── 02_output/ └── graficos/ └── boxplot.png ``` --- ### Definir directorio de trabajo `\(~\)` ``` r # Ruta para la carpeta donde vamos a guardar los archivos: path <- "D:/curso_r" # si estás usando Windows, nota que la ruta que copiaste está con las barras posicionadas en sentido contrario al ejemplo de arriba (\ en lugar de /). Es necesario corregirlas en el script antes de ejecutar el comando. setwd(path) # definir el directorio getwd() # este comando muestra cuál directorio está en uso dir() # visualizar los archivos y subcarpetas del directorio # guardar workspace ``` --- .center[ #### Versión de R instalada y directorio ] `\(~\)` <img src="index_files/figures/console.png" width="80%" style="display: block; margin: auto;" /> --- layout: false class: inverse, middle, center # Insertando datos --- ### Insertando datos `\(~\)` `\(~\)` .center[ ``` Matriz_Excel <- (filas = unidades muestrales, columnas = variables) ``` ] `\(~\)` .center[ ["datos_odonto.csv"](https://github.com/avilaf/curso_r/blob/cf708e2bb4116784de716f5ae1d58f9a141a256d/dados_odonto.csv#L1) `\(~\)` Dieta de dos poblaciones de *Odontophrynus asper* [(Machado et al., 2018)](https://avilaf.github.io/meus_artigos/Diet%20of%20Odontophrynus%20americanus%20in%20southern%20Atlantic%20Forest%20of%20Brazi.pdf) ] --- ``` r # Para importar datos en formato .csv que esté en tu directorio de trabajo: # datos_odonto <- read.csv("camino/para/archivo/dados_odonto.csv", header = TRUE) # Aquí vamos a abrir directamente del repositorio online de github: # install.packages("readr") library(readr) url <- "https://raw.githubusercontent.com/avilaf/curso_r/refs/heads/main/datos/dados_odonto.csv" datos_odonto <- readr::read_csv(url, locale = locale(encoding = "UTF-8")) ``` ``` ## Rows: 34 Columns: 36 ## ── Column specification ──────────────────────────────────────────────────────── ## Delimiter: "," ## chr (2): pop, sex ## dbl (34): ind, massa_g, crc_mm, gastropoda_n, gastropoda_v, araneae_n, arane... ## ## ℹ Use `spec()` to retrieve the full column specification for this data. ## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message. ``` --- ``` r datos_odonto ``` ``` ## # A tibble: 34 × 36 ## ind pop sex massa_g crc_mm gastropoda_n gastropoda_v araneae_n ## <dbl> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> ## 1 1 A M 7.8 37 0 0 0 ## 2 2 A M 11 38 0 0 0 ## 3 3 A F 12.3 45 0 0 0 ## 4 4 A M 16.2 48 0 0 0 ## 5 5 A M 14.5 44 0 0 0 ## 6 6 A F 23 55 0 0 0 ## 7 7 A M 11.6 44 0 0 0 ## 8 8 A M 12.7 45 0 0 1 ## 9 9 A M 12.1 43 0 0 0 ## 10 10 A M 14.6 44 0 0 0 ## # ℹ 24 more rows ## # ℹ 28 more variables: araneae_v <dbl>, coleoptera_n <dbl>, coleoptera_v <dbl>, ## # hymenoptera_n <dbl>, hymenoptera_v <dbl>, orthoptera_n <dbl>, ## # orthoptera_v <dbl>, lepidoptera_larva_n <dbl>, lepidoptera_larva_v <dbl>, ## # isopoda_n <dbl>, isopoda_v <dbl>, chilopoda_n <dbl>, chilopoda_v <dbl>, ## # diplopoda_n <dbl>, diplopoda_v <dbl>, lepidoptera_n <dbl>, ## # lepidoptera_v <dbl>, anelida_n <dbl>, anelida_v <dbl>, … ``` --- ``` r head(datos_odonto) ``` ``` ## # A tibble: 6 × 36 ## ind pop sex massa_g crc_mm gastropoda_n gastropoda_v araneae_n araneae_v ## <dbl> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> ## 1 1 A M 7.8 37 0 0 0 0 ## 2 2 A M 11 38 0 0 0 0 ## 3 3 A F 12.3 45 0 0 0 0 ## 4 4 A M 16.2 48 0 0 0 0 ## 5 5 A M 14.5 44 0 0 0 0 ## 6 6 A F 23 55 0 0 0 0 ## # ℹ 27 more variables: coleoptera_n <dbl>, coleoptera_v <dbl>, ## # hymenoptera_n <dbl>, hymenoptera_v <dbl>, orthoptera_n <dbl>, ## # orthoptera_v <dbl>, lepidoptera_larva_n <dbl>, lepidoptera_larva_v <dbl>, ## # isopoda_n <dbl>, isopoda_v <dbl>, chilopoda_n <dbl>, chilopoda_v <dbl>, ## # diplopoda_n <dbl>, diplopoda_v <dbl>, lepidoptera_n <dbl>, ## # lepidoptera_v <dbl>, anelida_n <dbl>, anelida_v <dbl>, dermaptera_n <dbl>, ## # dermaptera_v <dbl>, blattodea_n <dbl>, blattodea_v <dbl>, … ``` --- ``` r summary(datos_odonto) ``` ``` ## ind pop sex massa_g ## Min. : 1.00 Length:34 Length:34 Min. : 4.00 ## 1st Qu.: 9.25 Class :character Class :character 1st Qu.: 6.20 ## Median :17.50 Mode :character Mode :character Median :10.03 ## Mean :17.50 Mean :10.13 ## 3rd Qu.:25.75 3rd Qu.:12.60 ## Max. :34.00 Max. :23.00 ## crc_mm gastropoda_n gastropoda_v araneae_n ## Min. :23.00 Min. :0.0000 Min. : 0.00 Min. :0.0000 ## 1st Qu.:30.25 1st Qu.:0.0000 1st Qu.: 0.00 1st Qu.:0.0000 ## Median :38.50 Median :0.0000 Median : 0.00 Median :0.0000 ## Mean :37.88 Mean :0.2353 Mean : 23.03 Mean :0.1176 ## 3rd Qu.:44.00 3rd Qu.:0.0000 3rd Qu.: 0.00 3rd Qu.:0.0000 ## Max. :55.00 Max. :3.0000 Max. :408.00 Max. :1.0000 ## araneae_v coleoptera_n coleoptera_v hymenoptera_n ## Min. : 0.00 Min. :0.0000 Min. : 0.00 Min. :0.00000 ## 1st Qu.: 0.00 1st Qu.:0.0000 1st Qu.: 0.00 1st Qu.:0.00000 ## Median : 0.00 Median :0.0000 Median : 0.00 Median :0.00000 ## Mean : 41.79 Mean :0.4412 Mean : 32.32 Mean :0.08824 ## 3rd Qu.: 0.00 3rd Qu.:0.0000 3rd Qu.: 0.00 3rd Qu.:0.00000 ## Max. :960.00 Max. :4.0000 Max. :360.00 Max. :1.00000 ## hymenoptera_v orthoptera_n orthoptera_v lepidoptera_larva_n ## Min. : 0.000 Min. :0.00000 Min. : 0.00 Min. :0.0000 ## 1st Qu.: 0.000 1st Qu.:0.00000 1st Qu.: 0.00 1st Qu.:0.0000 ## Median : 0.000 Median :0.00000 Median : 0.00 Median :0.0000 ## Mean : 4.912 Mean :0.05882 Mean : 53.18 Mean :0.3824 ## 3rd Qu.: 0.000 3rd Qu.:0.00000 3rd Qu.: 0.00 3rd Qu.:0.0000 ## Max. :100.000 Max. :1.00000 Max. :1440.00 Max. :6.0000 ## lepidoptera_larva_v isopoda_n isopoda_v chilopoda_n ## Min. : 0.0 Min. :0.0000 Min. : 0.00 Min. :0.00000 ## 1st Qu.: 0.0 1st Qu.:0.0000 1st Qu.: 0.00 1st Qu.:0.00000 ## Median : 0.0 Median :0.0000 Median : 0.00 Median :0.00000 ## Mean : 113.4 Mean :0.5294 Mean : 18.65 Mean :0.05882 ## 3rd Qu.: 0.0 3rd Qu.:0.0000 3rd Qu.: 0.00 3rd Qu.:0.00000 ## Max. :2395.0 Max. :5.0000 Max. :294.00 Max. :2.00000 ## chilopoda_v diplopoda_n diplopoda_v lepidoptera_n ## Min. : 0.000 Min. :0.00000 Min. : 0.00 Min. :0.00000 ## 1st Qu.: 0.000 1st Qu.:0.00000 1st Qu.: 0.00 1st Qu.:0.00000 ## Median : 0.000 Median :0.00000 Median : 0.00 Median :0.00000 ## Mean : 1.059 Mean :0.05882 Mean : 13.65 Mean :0.05882 ## 3rd Qu.: 0.000 3rd Qu.:0.00000 3rd Qu.: 0.00 3rd Qu.:0.00000 ## Max. :36.000 Max. :1.00000 Max. :400.00 Max. :1.00000 ## lepidoptera_v anelida_n anelida_v dermaptera_n dermaptera_v ## Min. : 0.000 Min. :0.0000 Min. : 0.00 Min. :0 Min. :0 ## 1st Qu.: 0.000 1st Qu.:0.0000 1st Qu.: 0.00 1st Qu.:0 1st Qu.:0 ## Median : 0.000 Median :0.0000 Median : 0.00 Median :0 Median :0 ## Mean : 1.765 Mean :0.2353 Mean : 13.76 Mean :0 Mean :0 ## 3rd Qu.: 0.000 3rd Qu.:0.0000 3rd Qu.: 0.00 3rd Qu.:0 3rd Qu.:0 ## Max. :30.000 Max. :7.0000 Max. :300.00 Max. :0 Max. :0 ## blattodea_n blattodea_v neoroptera_n neoroptera_v ## Min. :0.00000 Min. : 0.00 Min. :0.00000 Min. : 0.000 ## 1st Qu.:0.00000 1st Qu.: 0.00 1st Qu.:0.00000 1st Qu.: 0.000 ## Median :0.00000 Median : 0.00 Median :0.00000 Median : 0.000 ## Mean :0.02941 Mean : 11.12 Mean :0.02941 Mean : 2.941 ## 3rd Qu.:0.00000 3rd Qu.: 0.00 3rd Qu.:0.00000 3rd Qu.: 0.000 ## Max. :1.00000 Max. :378.00 Max. :1.00000 Max. :100.000 ## otros n_total vol_total ## Min. : 0.0 Min. : 0.000 Min. : 0.00 ## 1st Qu.: 0.0 1st Qu.: 0.000 1st Qu.: 89.25 ## Median : 40.0 Median : 1.000 Median : 188.50 ## Mean :125.4 Mean : 2.324 Mean : 457.03 ## 3rd Qu.:142.0 3rd Qu.: 3.000 3rd Qu.: 551.25 ## Max. :962.0 Max. :13.000 Max. :2963.00 ``` --- `\(~\)` .pull-left[ ``` r # podemos aislar esos datos y hacer # muchas cosas con la información. # Por ejemplo un gráfico: # install.packages("ggplot2") library(ggplot2) theme_set(theme_bw()) ggplot(data = datos_odonto, aes(x = crc_mm, y = massa_g)) + geom_point(color = "gold3") + geom_smooth(method = "lm", se = F, color = "firebrick") + labs(x = "Largo hocico-cloaca (mm)", y = "Masa (g)") ``` ] .pull-right[ 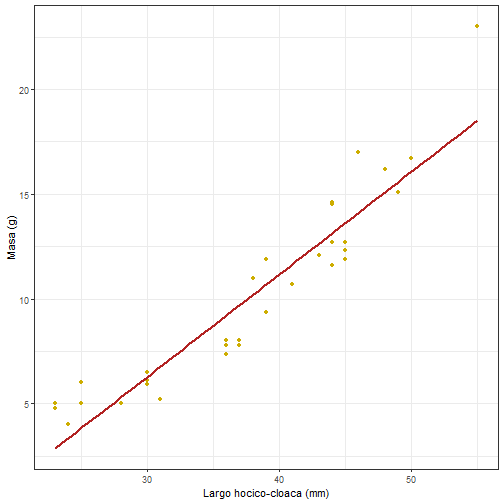<!-- --> ] --- layout: false class: inverse, middle, center # Convertir datos --- ### Convertir datos Funciones: **as.numeric()**, **as.character()**, **as.logical()** etc. ``` r # Ejemplo: a <- "42" # atribuye el valor de "42" al objeto "a" class(a) # Retorna "character" ``` ``` ## [1] "character" ``` ``` r a # Nota que el número está escrito entre comillas. ``` ``` ## [1] "42" ``` ``` r # Este formato en el lenguaje R significa que este número está almacenado como un "character" o "string", o sea, como texto. ``` --- ### Texto para número `\(~\)` ``` r b <- as.numeric(a) # Convierte el string "42" para número 42 b ``` ``` ## [1] 42 ``` ``` r class(b) # Retorna "numeric" ``` ``` ## [1] "numeric" ``` --- ### Ejemplo `\(~\)` `\(~\)` ``` r # con nuestros datos de anfibios ind <- c(datos_odonto$ind) ind ``` ``` ## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 ## [26] 26 27 28 29 30 31 32 33 34 ``` ``` r class(ind) # R interpreta esos datos como valores numéricos ``` ``` ## [1] "numeric" ``` --- ## Cómo "character" `\(~\)` `\(~\)` ``` r ind_string <- as.character(ind) # queremos que sepa que esos números son los "nombres" de nuestras muestras ind_string ``` ``` ## [1] "1" "2" "3" "4" "5" "6" "7" "8" "9" "10" "11" "12" "13" "14" "15" ## [16] "16" "17" "18" "19" "20" "21" "22" "23" "24" "25" "26" "27" "28" "29" "30" ## [31] "31" "32" "33" "34" ``` ``` r class(ind_string) ``` ``` ## [1] "character" ``` --- layout: false class: inverse, middle, center # Paquetes --- ### Paquetes `\(~\)` `\(~\)` .center[ **Funciones básicas** implementadas sin bajar y cargar bibliotecas adicionales **Paquetes:** cajas de herramientas que facilitan analizar datos, hacer gráficos, mapas... que **extienden las funcionalidades** básicas ] `\(~\)` .center[ ``` pauquetes <- conjuntos_organizados_de(funciones, datos, documentación) ``` ] `\(~\)` --- #### En RStudio: **"Packages"** : - los **paquetes cargados** estarán señalizados - puedes cargar los paquetes <img src="index_files/figures/rstudio_packages.png" width="40%" style="display: block; margin: auto;" /> `\(~\)` .center[ *Lo ideal es tener el comando para cargar el paquete detallado siempre en tu script* así tendrás las etapas de tu proyecto **automatizadas** y no correrás el riesgo de olvidar algún paquete importante. ] --- ### Ejemplo ``` r # Creando un data frame de ejemplo datos <- data.frame(idade = c(25, 30, NA, 40, 35)) # Función base mean() mean(datos) ``` ``` ## Warning in mean.default(datos): argumento não é numérico nem lógico: retornando ## NA ``` ``` ## [1] NA ``` -- #### Qué paso? Respuesta = **vector vacío "[NA]"** Esto ocurre porque la función base, por default, no calcula promedio cuando uno de los argumentos no sea numérico. --- .center[ ### Qué hacemos? ] `\(~\)` `\(~\)` `\(~\)` <img src="index_files/figures/kermit_sad.png" width="40%" style="display: block; margin: auto;" /> --- ### Opción 1: `\(~\)` Implementar una función nueva: ``` r # Función creada por el usuario para calcular promedio ignorando NAs minha_media <- function(x) { x_sim_na <- x[!is.na(x)] suma <- sum(x_sim_na) n <- length(x_sim_na) media <- suma / n return(media) } # Aplicando la función minha_media(datos$idade) ``` ``` ## [1] 32.5 ``` --- ### Opción 2: `\(~\)` `\(~\)` Paquete **"dplyr"**: editar datos y extraer mucha información. .pull-left[ ``` r # Instalar paquete # install.packages("dplyr") # Cargar el paquete library(dplyr) ``` ] .pull-right[ ``` r # Usando la función summarise de dplyr datos %>% summarise(media_idade = mean(idade, na.rm = TRUE)) ``` ``` ## media_idade ## 1 32.5 ``` ] --- ### Opción 3: `\(~\)` `\(~\)` Corregir la función `base::mean` ``` r mean(datos$idade, na.rm = TRUE) ``` ``` ## [1] 32.5 ``` `\(~\)` `\(~\)` -- #### Cómo sabemos? --- layout: false class: inverse, middle, center # Buscando ayuda --- `\(~\)` **Otros softwares estadísticos pagos:** ayuda = asistencia del provedor -- **R:** software libre = sin asistencia? -- `\(~\)` <img src="index_files/figures/y-ahora-quien-podra-defenderme.jpg" width="30%" style="display: block; margin: auto;" /> --- ### Comunidad R **Grande, activa y colaborativa:** - Páginas web - Grupos de email - Foros - Tutoriales <img src="index_files/figures/no-contavan-con-mi-astucia.jpg" width="30%" style="display: block; margin: auto;" /> --- ## Documentación de los paquetes `\(~\)` `\(~\)` ``` r # Supongamos que queremos saber más sobre la función "plot" implementada en la base del propio R ?base::plot # o help(plot) ``` --- ### Ejemplo .pull-left[ `\(~\)` `\(~\)` ``` r # copiamos la información disponible en el #"Help", la ejecutamos y analizamos # cómo se comporta el código: require(graphics) # for plot methods plot(cars) ``` ] .pull-right[ 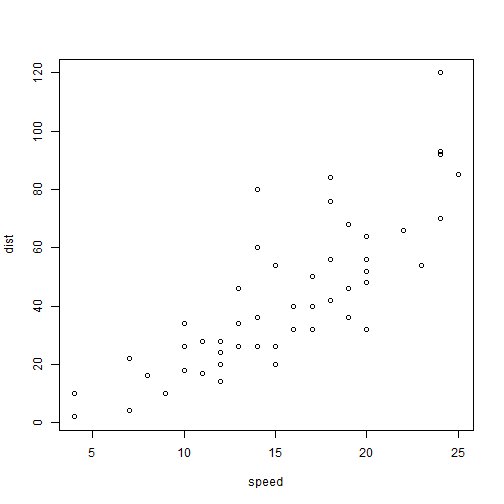<!-- --> ] --- ### Ahora adaptamos para nuestro contexto: .pull-left[ `\(~\)` ``` r # Organizamos los datos datos_plot <- data.frame(LHC = datos_odonto$crc_mm, Masa = datos_odonto$massa_g) plot(datos_plot) ``` ] .pull-right[ 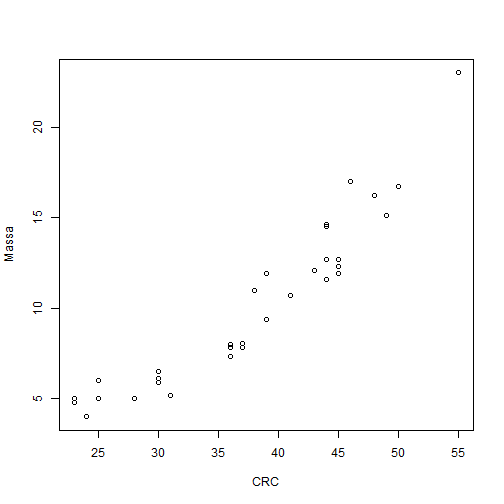<!-- --> ] --- ### Línea de tendencia .pull-left[ `\(~\)` ``` r # la función para calcular y graficar # la línea de tendencia es un poco más # compleja, vamos a buscar ayuda #?abline # también podemos alterar parámetros # estéticos del gráfico plot(Masa ~ LHC, data = datos_plot, col = 7, pch = 16) abline(lm(Masa ~ LHC, data = datos_plot), col = 2) # Nota que este comando contiene el gráfico de la línea y la ecuación de la recta, dos funciones. ¿Puedes identificar esos elementos en el código? ``` ] .pull-right[ 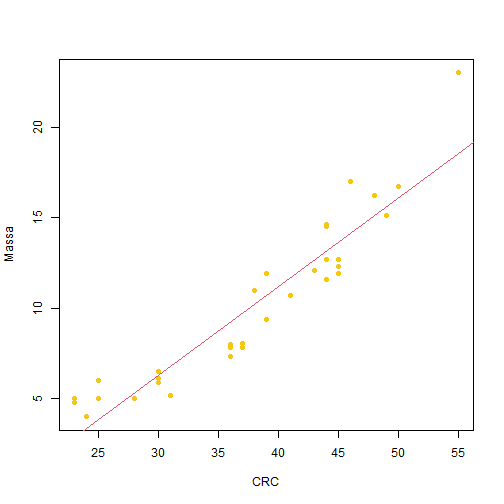<!-- --> ] --- ### Resumen: `\(~\)` Al final conseguimos ejecutar varias tareas: `\(~\)` - Gráfico de dispersión simple; - Ecuación de la recta; - Gráfico estilizado con la línea de tendencia. <img src="index_files/figures/sapo_pc.jpg" width="50%" style="display: block; margin: auto;" /> --- layout: false class: inverse, middle, center # Exportando datos y figuras --- ### Datos Es natural que, una vez hecha alguna transformación o cálculo con tus datos brutos, quieras **exportarlos en un archivo que pueda ser visualizado y manipulado en otros softwares** fuera del ambiente R. Esta es una tarea simple, que podemos hacer en el propio script de la siguiente forma: `\(~\)` ``` r # supongamos que necesitas exportar el objeto que creamos para # el gráfico en un archivo .csv que pueda ser abierto con Excel datos_plot # este objecto R class(datos_plot) write.csv(datos_plot,"~/01_datos/datos_plot.csv") ``` `\(~\)` Puedes verificar en tu directorio si el archivo fue guardado y cómo fue guardado. --- ## Figuras De la misma forma podemos exportar para nuestro directorio los **gráficos o mapas** ``` r # Son 3 pasos # 1- Crear la imagen jpg('~/02_resultados/grafico_odonto.jpg') # 2- Crear el gráfico plot(Massa ~ CRC, data = datos_plot, col = 7, pch = 16) abline(lm(Massa ~ CRC, data = datos_plot), col = 2) # 3- Cerrar el archivo dev.off() ``` Así como hicimos con los datos, puedes verificar en tu directorio si la figura fue exportada y cómo fue guardada. --- ### Exportar gráficos con RStudio Otra opción igualmente práctica para guardar una figura a partir de RStudio es guardar haciendo clic en el **botón Export**, en la parte superior de la ventana **Plots**: <img src="index_files/figures/grafico.png" width="50%" style="display: block; margin: auto;" /> --- layout: false class: inverse, middle, center # Buenas prácticas escribiendo comandos --- ### Buenas prácticas escribiendo comandos `\(~\)` .center[ El intérprete de R es bastante eficiente y **no exige que tengamos gran afinidad con lenguaje de programación** para utilizarlo. `\(~\)` Esto facilita mucho nuestro aprendizaje y nuestro trabajo. ] `\(~\)` `\(~\)` <img src="index_files/figures/sapo.jpg" width="25%" style="display: block; margin: auto;" /> --- ### Ejemplo **R no tiene en cuenta:** espaciado entre caracteres y indentación (organizar el código en párrafos) ``` r # R interpreta igualmente los dos codigos: # 1- con espaciado y indentación bonito <- c("Ololygon", "Scinax", "Boana") bonito ``` ``` ## [1] "Ololygon" "Scinax" "Boana" ``` ``` r # 2- sin espaciado ni identación feo<-c("Ololygon","Scinax","Boana") feo ``` ``` ## [1] "Ololygon" "Scinax" "Boana" ``` --- ### Importante mantener el material legible `\(~\)` `\(~\)` .center[ #### no es porque el intérprete de la máquina puede leer, que otra persona que va a replicar tu código (o tú mismo/a en el futuro) va a descifrar la lógica de tu proyecto ] -- `\(~\)` `\(~\)` ### O sea... .center[ Tener en cuenta la **legibilidad del código** = trabajo replicable y eficiente ] --- layout: false class: inverse, middle, center # Ejemplo de *Python* --- ### Ejemplo de *Python* `\(~\)` .center[ Lenguaje de programación que también es ampliamente utilizado en análisis de datos, que posee una comunidad gigante y muy activa Para ese lenguaje existe una serie de recomendaciones bien humoradas llamadas el [Zen of Python](https://pt.wikipedia.org/wiki/Zen_of_Python) (**principios de diseño**) ] <img src="index_files/figures/Python-Logo.png" width="30%" style="display: block; margin: auto;" /> --- ## Zen of Python `\(~\)` - **Hermoso** es mejor que feo. ``` r # Feo f <- function(x) if(x > 0) x + 1 else x - 1 # Hermoso ajusta_valor <- function(x) { if (x > 0) { return(x + 1) } else { return(x - 1) } } ``` --- `\(~\)` `\(~\)` `\(~\)` `\(~\)` - **Explícito** es mejor que implícito. ``` r # Implícito (puede causar confusión si muchas bibliotecas son cargadas) library(tidyverse) filter(data) # Puede ser ambiguo (conflicto con 'stats::filter') # Explícito dplyr::filter(data) # Claro de dónde viene la función ``` --- `\(~\)` `\(~\)` `\(~\)` `\(~\)` - **Simple** es mejor que complejo. - **Complejo** es mejor que complicado. ``` r # Complejo result <- sapply(1:10, function(x) if(x %% 2 == 0 && x > 4) x^2 else NULL) # Simple result <- c() for (x in 1:10) { if (x %% 2 == 0 && x > 4) { result <- c(result, x^2) } } ``` --- `\(~\)` `\(~\)` `\(~\)` `\(~\)` - **E s p a r s o** es mejor que *denso*. - **Legibilidad** cuenta. ``` r # Menos legible x<-1;y<-2;z<-x+y # Más legible x <- 1 y <- 2 z <- x + y ``` -- - Si la implementación es **difícil de explicar, es una mala idea** - Si la implementación es **fácil de explicar, puede ser una buena idea** --- layout: false class: inverse, middle, center # Organizar los scripts --- ### Organizar los scripts #### Sugestión - Un **encabezado** con: título, descripción de los análisis, tu nombre, la fecha del análisis y otras informaciones pertinentes -- - Un sector para **bajar y cargar los paquetes** necesarios durante el análisis -- - Un sector para asignar el **directorio de trabajo** -- - Otro para **cargar los datos** -- - El desarrollo del **análisis** -- - La finalización de los análisis con la exportación de los **resultados** -- - Finalizar con las **referencias** de los paquetes y de R para reportar en tu publicación. --- ### Ejemplo pt. 1 `\(~\)` - Un **encabezado** con: título, descripción de los análisis, tu nombre, la fecha del análisis y otras informaciones pertinentes - Un sector para **bajar y cargar los paquetes** necesarios durante el análisis ``` r # Plotando um mapa simples com o pacote "mapdata" # Example worksheet by Fernanda Rodrigues de Avila # R version 4.5.0 # Paquete ----------------------------------------------------------------- # install.packages("mapdata") # instalar library(mapdata) # cargar ``` --- ### Ejemplo pt. 2 `\(~\)` - Un sector para **cargar los datos** - El desarrollo del **análisis** ``` r # Cargando datos ---------------------------------------------------------- RegHR <- map("worldHires", namesonly = TRUE, plot = FALSE) # Mapa -------------------------------------------------------------------- mapa <- map('worldHires', fill = TRUE, col = terrain.colors(6), bg = "#CCEEFF", region = RegHR[-grep("Antarctica", RegHR)]) mapa # ver el mapa ``` --- ### Ejemplo pt. 3 `\(~\)` - La finalización de los análisis con la exportación de los **resultados** ``` r # Exportar mapa -------------------------------------------------------- # 1- Crear la imagen jpg('~/mapa_ejemplo.jpg') # local para salvar # 2- Repetir mapa mapa # 3- Cerrar el archivo dev.off() ``` --- ### Ejemplo pt. 4 (final) `\(~\)` - Finalizar con las **referencias** de los paquetes y de R para reportar en tu publicación ``` r # Referencias ---------------------------------------------------------- citation() # R RStudio.Version() # RStudio citation("mapdata") # Paquete "mapdata" ###############################_fin_#################################### # R version 4.5.0 (2025-04-11 ucrt) # Dra. Fernanda Rodrigues de Avila # <https://avilaf.github.io/> # fernandar.avila@gmail.com ``` --- layout: false class: inverse, middle, center # Pseudocódigo --- ### Pseudocódigo `\(~\)` .center[ Útil cuando aún **no conocemos muchas funciones** o formas de interactuar con un lenguaje de programación **Pseudocódigo**: un esquema lógico, en nuestra lengua materna, que seguimos para realizar nuestro trabajo en R (o en otro lenguaje) ] `\(~\)` -- .center[ #### Importante Como el lenguaje **R es muy simple**, probablemente no lo vas a utilizar por mucho tiempo Pero puede ser una **herramienta didáctica** interesante para iniciar tu jornada en este nuevo lenguaje ] --- ### Ejemplo pt. 1 Has hecho campos nocturnos en algunos sitios reproductivos y muestreado las **comunidades de anfibios** -- Después de los campos tabulaste los datos y generaste el siguiente dataframe en tu archivo de Excel: <img src="index_files/figures/excel.png" width="50%" style="display: block; margin: auto;" /> -- `\(~\)` Abundancia de anfibios -- Diferentes especies y comunidades -- Días de muestreo -- Tipo de hábitat ("agr" = agricultura y "nat" = hábitat natural) -- La columna "local" informa cuatro lugares de colecta ("a", "b", "c" y "d") --- ### Ejemplo pt. 2 Tu objetivo es calcular la **abundancia total** en cada sitio de colecta en cada noche de muestreo -- Quieres hacer esto en R, pero aún no estás familiarizado/a con el lenguaje -- `\(~\)` Puedes utilizar el pseudocódigo para organizar tus ideas antes de escribir el código real: ``` r # En tu script, usa comentarios (#) para organizar el pseudocódigo y escribe las etapas # datos <- importar (archivo.csv) # abundancias <- recortar variables respuesta (datos) # suma <- sumar las filas (abundancias) # datos_completos <- juntar (datos, abundancias) ``` --- ### Ejemplo pt. 3 ``` r # ahora que tenemos un camino que parece simple y puede funcionar, buscando las funciones correctas puedes escribir los comandos # datos <- importar (archivo.csv) datos <- read.table(header = TRUE, text = "dia habitat local Dendropsophus_sanborni Dendropsophus_minutus Pseudopaludicola_falcipes Boana_pulchella Leptodactylus_gracilis Scinax_fuscovarius 1 agr a 10 0 15 1 3 2 1 agr b 6 11 20 7 1 9 1 nat c 13 3 18 4 6 5 1 nat d 9 14 23 10 4 12 2 agr a 8 0 13 0 1 0 2 agr b 4 9 18 5 0 7 2 nat c 11 1 16 2 4 3 2 nat d 7 12 21 8 2 10 3 agr a 11 1 16 2 4 3 3 agr b 7 12 21 8 2 10 3 nat c 14 4 19 5 7 6 3 nat d 10 15 24 11 5 13 4 agr a 9 0 14 0 2 1 4 agr b 5 10 19 6 0 8 4 nat c 13 3 18 4 6 5 4 nat d 9 14 23 10 4 12 ") ``` --- ### Ejemplo pt. 4 (final) ``` r # abundancias <- recortar variables respuesta (datos) abundancias <- datos[1:16,4:9] # suma <- sumar las filas (abundancias) suma <- as.vector(rowSums(abundancias)) # datos_completos <- juntar (datos, abundancias) datos_completos <- cbind(datos, suma) datos_completos$suma # la última columna de este dataframe almacena la suma de las abundancias ``` ``` ## [1] 31 54 49 72 22 43 37 60 37 60 55 78 26 48 49 72 ``` -- .pull-left[ #### Simple? ] .pull-right[ <img src="index_files/figures/kermit_think.png" width="40%" style="display: block; margin: auto;" /> ] --- ### Más allá de los números... Vamos a hacer un gráfico con las abundancias, por diversión: .pull-left[ `\(~\)` ``` r # Gráfico boxplot(suma ~ habitat, data = datos_completos, col = c(7, 3) ) # Abundancias totales por habitat ``` Parece que los valores de abundancia no son iguales entre los tratamientos ] .pull-right[ <img src="curso_r_slides_clase_2_files/figure-html/fugura_bound-out-1.png" width="70%" /> ] --- ### Vamos a jugar un poco más Podemos hacer una prueba rápida para descubrir si ese patrón visual tiene significancia estadística ``` r # Para comparar muestras de dos tratamientos # con variables respuesta cuantitativas (como es nuestro caso) # podemos usar una Teste T de Student (paramétrico) # o Mann-Whitney (no paramétrica). # Vamos a probar los supuestos de la Prueba T antes de calcular: # probar la normalidad con Shapiro-Wilk # H0: los datos de la muestra provienen de una población con distribución normal shapiro.test(x = datos_completos$suma) ``` ``` ## ## Shapiro-Wilk normality test ## ## data: datos_completos$suma ## W = 0.97118, p-value = 0.8573 ``` ``` r # p > 0,05: no rechazamos la premisa de normalidad de los datos (H0) ``` --- ### Graficar la distribuición .pull-left[ ``` r # Graficar la probabilidad de # la distribución normal: qqnorm(datos_completos$suma, pch = 1, frame = FALSE) # datos qqline(datos_completos$suma, col = "steelblue", lwd = 2) # recta # parece que los datos se # ajustan bien a la normalidad ``` ] .pull-right[ <img src="curso_r_slides_clase_2_files/figure-html/distribuicion-out-1.png" width="70%" /> ] --- ### Homogeneidad de las varianzas ``` r # Paquete: # install.packages("car") # library(car) # Levene's test # H0: las varianzas de las poblaciones son iguales car::leveneTest(suma ~ habitat, data = datos_completos) ``` ``` ## Warning in leveneTest.default(y = y, group = group, ...): group coerced to ## factor. ``` ``` ## Levene's Test for Homogeneity of Variance (center = median) ## Df F value Pr(>F) ## group 1 0.0121 0.9138 ## 14 ``` ``` r # p > 0,05: no rechazamos la premisa de homogeneidad de las varianzas (H0) ``` --- ### Teste T ``` r # Ahora la prueba teste_t <- t.test(suma ~ habitat, data = datos_completos, var.equal = T) teste_t ``` ``` ## ## Two Sample t-test ## ## data: suma by habitat ## t = -2.729, df = 14, p-value = 0.0163 ## alternative hypothesis: true difference in means between group agr and group nat is not equal to 0 ## 95 percent confidence interval: ## -33.709275 -4.040725 ## sample estimates: ## mean in group agr mean in group nat ## 40.125 59.000 ``` --- layout: false class: inverse, middle, center # ¿Qué es un Pipe? --- # ¿Qué es un Pipe? **pipe** <- operador para encadenar operaciones de manera más legible En lugar de **anidar funciones** o crear múltiples **objetos intermedios** "pasar" el resultado de una operación directamente a la siguiente <div class="figure" style="text-align: center"> <img src="index_files/figures/MagrittePipe.jpg" alt="The Treachery of Images, 1929, Rene Magritte" width="40%" /> <p class="caption">The Treachery of Images, 1929, Rene Magritte</p> </div> --- ## Los dos tipos de pipes en R - **Pipe nativo** `|>`: Disponible en R base desde la versión 4.1.0 - **Pipe de magrittr** `%>%`: Del paquete magrittr (incluido en tidyverse) --- ## Problema: Código sin pipes Imagina que queremos procesar un conjunto de datos paso a paso: ``` r # CÁLCULO DE CONDICIÓN CORPORAL EN ANFIBIOS library(dplyr) # Datos de ejemplo datos <- data.frame( ind = c("1", "2", "3", "4", "5"), sp = c("a", "a", "b", "b", "b"), masa = c(8, 11, 12, 16, 14), lhc = c(37, 38, 45, 48, 44) ) print(datos) ``` ``` ## ind sp masa lhc ## 1 1 a 8 37 ## 2 2 a 11 38 ## 3 3 b 12 45 ## 4 4 b 16 48 ## 5 5 b 14 44 ``` --- ## Método tradicional (anidado) - DIFÍCIL DE LEER ``` r calcular_icc_anidado <- function(df) { df$icc_anidado <- round((df$masa / (df$lhc^3)) * 10000, digits = 4) return(df) } # Aplicando el método anidado resultado_anidado <- calcular_icc_anidado(datos) print(resultado_anidado[, c("ind", "sp", "masa", "lhc", "icc_anidado")]) ``` ``` ## ind sp masa lhc icc_anidado ## 1 1 a 8 37 1.5794 ## 2 2 a 11 38 2.0047 ## 3 3 b 12 45 1.3169 ## 4 4 b 16 48 1.4468 ## 5 5 b 14 44 1.6435 ``` --- ## Método con objetos intermedios - MUCHAS VARIABLES ``` r datos2 <- data.frame( ind = c("1", "2", "3", "4", "5"), sp = c("a", "a", "b", "b", "b"), masa = c(8, 11, 12, 16, 14), lhc = c(37, 38, 45, 48, 44) ) ``` --- ## Método con objetos intermedios pt. 2 ``` r masa <- datos2$masa lhc_cubo <- c(datos2$lhc^3) razon <- c(masa / lhc_cubo) escala <- c(razon * 10000) redondear_resultado <- round(escala, digits = 4) datos2$icc_intermedios <- redondear_resultado print(datos2) ``` ``` ## ind sp masa lhc icc_intermedios ## 1 1 a 8 37 1.5794 ## 2 2 a 11 38 2.0047 ## 3 3 b 12 45 1.3169 ## 4 4 b 16 48 1.4468 ## 5 5 b 14 44 1.6435 ``` --- ## Solución: Usando pipes ### Con el pipe de magrittr (`%>%`) ``` r library(dplyr) library(magrittr) # Método con %>% - FÁCIL DE LEER datos3 <- data.frame( ind = c("1", "2", "3", "4", "5"), sp = c("a", "a", "b", "b", "b"), masa = c(8, 11, 12, 16, 14), lhc = c(37, 38, 45, 48, 44) ) ``` --- ### Con el pipe de magrittr (`%>%`) pt. 2 ``` r resultado_pipe <- datos3 %>% mutate( icc_pipe = masa %>% divide_by(lhc^3) %>% multiply_by(10000) %>% round(digits = 4) ) print(resultado_pipe) ``` ``` ## ind sp masa lhc icc_pipe ## 1 1 a 8 37 1.5794 ## 2 2 a 11 38 2.0047 ## 3 3 b 12 45 1.3169 ## 4 4 b 16 48 1.4468 ## 5 5 b 14 44 1.6435 ``` --- ## Con el pipe nativo (`|>`) ``` r # Método con |> - TAMBIÉN FÁCIL DE LEER datos4 <- data.frame( ind = c("1", "2", "3", "4", "5"), sp = c("a", "a", "b", "b", "b"), masa = c(8, 11, 12, 16, 14), lhc = c(37, 38, 45, 48, 44) ) ``` --- ## Con el pipe nativo (`|>`) pt. 2 ``` r resultado_nativo <- datos4 |> mutate( icc_pipe = masa |> divide_by(lhc^3) |> multiply_by(10000) |> round(digits = 4) ) print(resultado_nativo) ``` ``` ## ind sp masa lhc icc_pipe ## 1 1 a 8 37 1.5794 ## 2 2 a 11 38 2.0047 ## 3 3 b 12 45 1.3169 ## 4 4 b 16 48 1.4468 ## 5 5 b 14 44 1.6435 ``` --- ## ¿Cómo funcionan los pipes? Los pipes toman el resultado del lado izquierdo y lo pasan como **primer argumento** a la función del lado derecho: ``` r # Sin pipe sqrt(16) ``` ``` ## [1] 4 ``` ``` r # Con %>% 16 %>% sqrt() ``` ``` ## [1] 4 ``` ``` r # Con |> 16 |> sqrt() ``` ``` ## [1] 4 ``` --- ## Ejemplo paso a paso ``` r # Creamos un vector numeros <- c(1, 4, 9, 16, 25) # Sin pipes resultado1 <- sqrt(sum(numeros)) print(paste("Sin pipes:", resultado1)) ``` ``` ## [1] "Sin pipes: 7.41619848709566" ``` --- ## Ejemplo paso a paso pt. 2 ``` r # Con %>% resultado2 <- numeros %>% sum() %>% sqrt() print(paste("Con %>%:", resultado2)) ``` ``` ## [1] "Con %>%: 7.41619848709566" ``` --- ## Ejemplo paso a paso pt. 3 ``` r # Con |> resultado3 <- numeros |> sum() |> sqrt() print(paste("Con |>:", resultado3)) ``` ``` ## [1] "Con |>: 7.41619848709566" ``` --- ## Ventajas de usar pipes 1. **Legibilidad**: El código se lee de izquierda a derecha, de arriba abajo 2. **Menos objetos intermedios**: No necesitamos crear variables temporales 3. **Menos errores**: Evitamos problemas con paréntesis anidados --- ## Consejos: ## ✅ **Buenas prácticas** - Usa una línea por operación - Identa correctamente el código - Combina pipes con funciones de `dplyr` para análisis de datos ## ❌ **Evita** - Pipes muy largos (más de 8-10 operaciones) - Mezclar `%>%` y `|>` en el mismo código sin razón --- ### Ejercicios `\(~\)` .center[ Ahora que sabes insertar, trabajar y exportar datos, buscar ayuda puedes aventurarte un poco y practicar algunos comandos con los ejercicios de esta etapa `ejercicios_2.Rmd` ] <img src="index_files/figures/kermit_tranqui.png" width="50%" style="display: block; margin: auto 0 auto auto;" /> --- `\(~\)` `\(~\)` <div class="figure" style="text-align: center"> <img src="index_files/figures/site.png" alt="avilaf.github.io/esp" width="70%" /> <p class="caption">avilaf.github.io/esp</p> </div> <p style="text-align:center;">fernandar.avila@gmail.com</p> <p style="text-align:center;">Lab. de Herpetología - FCIEN, UDELAR</p>